Charting mit ggplot2
ggplot2 ist ein tolles Package, um vielseitige Charts zu erstellen. Heute geht es darum, solche Charts mit dem ggplot2-Package in R zu erstellen. Wir schauen uns die Basis-Syntax an, erstellen unter anderem Histogramme, Boxplots, Lineplots oder Scatterplots und machen uns mit einigen Extras vertraut. Vorab sei gesagt: ggplot ist umfangreich und liefert eine Menge Funktionen und Parameter, sodass ich hier nur auf die Grundlagen eingehe. Es gibt einfach zu viele Möglichkeiten, die man alle in einem Post wohl kaum behandeln kann (z.B. Texte mit Pfeilen in unterschiedlichen Farben und Größen um 90 Grad rotiert auf verschiedene Subcharts plotten…). Also fangen wir an.
Das ggplot2-Package
ggplot2 laden
Bevor Du einen ggplot-Chart erstellen kannst, musst du die entsprechende Library laden. Auch wenn ich hier meistens von ggplot sprechen werde, heißt das Package ggplot2. Also als erstes library(ggplot2) eingeben (bzw. wenn noch nicht vorhanden, vorab install.packages("ggplot2")). Hier sei noch erwähnt, dass ggplot zum Tidyverse gehört (eine Sammlung an Paketen, die in ihrer Syntax ähnlich sind und sich super ergänzen). Somit würde ein install.packages("tidyverse") das Installieren von ggplot2 einschließen.
Die ggplot2-Syntax
ggplot basiert auf der Grammar of Graphics – ein System, das einen strukturierten Ansatz zur Gestaltung von Grafiken bzw. Visualisierungen liefert. So wird hier z.B. logisch getrennt zwischen den zugrundeliegenden Daten, deren Zuordnung zu entsprechenden Achsen (z.B. X oder Y), der Darstellung der Daten (Linien, Balken, Punkte, …) und weiteren Aspekten. Das werden wir gleich auch noch in der Syntax sehen; da wir hier weniger theoretisch als praktisch arbeiten wollen, springen wir jetzt gleich zu den ersten Zeilen Code.
Simulationsdaten generieren
Bevor wir unseren ersten Plot erstellen können, brauchen wir allerdings noch Daten. Ich benutze hier das data.table Package (siehe hier für eine Einführung) und einige Zufallsfunktionen, um synthetische Daten zu erstellen. Mit set.seed(2020) ist sichergestellt, dass die Zufallsfunktionen immer die selben Ergebnisse hervorbringen. Die resultierende Tabelle besteht aus drei Spalten: eine für die Gruppenzugehörigkeit (Group: entweder „A“, „B“, oder „C“) und zwei numerische (X und Y). Ich habe die Daten so erstellt, dass bestimmte Abhängigkeiten bestehen, die wir hoffentlich gleich in den Charts erkennen werden…
N <- 500
dt <- data.table(
Group = LETTERS[rbinom(N, 2, rep(1/3, 3)) + 1],
X = rnorm(N, 50, 5)
)
dt[X <= 45, Group := LETTERS[rbinom(.N, 1, 0.4) + 1]]
dt[X > 55, Group := LETTERS[rbinom(.N, 1, 0.6) + 2]]
dt[Group=="A", Y := 10 + 1.25*X + rnorm(.N, 0, 10)]
dt[Group=="B", Y := 10 + 1.35*X + rnorm(.N, 0, 10)]
dt[Group=="C", Y := X - 50 + 1.45*X + rnorm(.N, 0, 10)]Histogramme mit ggplot2
Unser erster Plot
Unser erster Plot ist einer der einfachsten, da er nur die Verteilung einer einzigen numerischen Variable darstellt – ein Histogramm. Wir können dieses in einer Zeile erstellen: ggplot(dt, aes(X)) + geom_histogram(bins=20). Hier sehen wir auch gleich die Besonderheit der ggplot2-Syntax: Mit ggplot(dt, aes(X)) erstellen wir den Plot, übergeben die Daten dt und definieren die sog. aesthetics, die Zuordnung (das mapping) von Variablen auf visuelle Aspekte. In unserem Fall wird lediglich angegeben, dass wir Variable X verwenden. Mit geom_histogram(bins=20) wird festgelegt, was für ein geometrisches Objekt (= hier: was für ein Charttyp) geplottet werden soll und welche weiteren Eigenschaften es beseitzt (z.B. sagt bins=20 an, dass das Histogramm aus 20 Klassen, oder bins, bestehen soll). Hier sehen wir das Resultat:
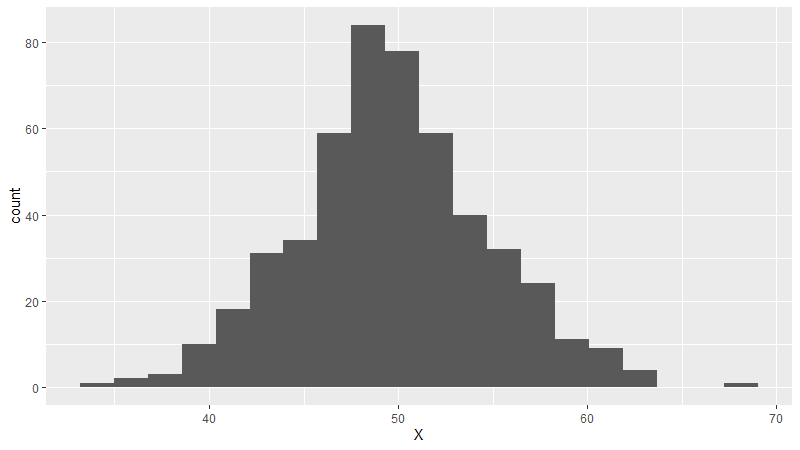
Weitere Attribute für Histogramme
geom_histogram ermöglicht auch weitere Einstellungen. Ein Beispiel: ggplot(dt) + geom_histogram(aes(y=X), binwidth=0.5, fill="darkgoldenrod"). Auch hier ein paar Erklärungen: Zum einen sehen wir, dass wir das aes(…) nicht mehr im ersten Teil enthalten haben – in diesem Fall bezieht es sich direkt auf das geometrische Objekt (geom_histogram). Auch haben wir hier ein explizites y=X in den Klammern von aes. Entsprechend wird Variable X hier auf die y-Achse gemapped. Mit binwidth=0.5 wird im Gegensatz zu vorher nicht die Anzahl der Klassen, sondern die Breite der Klassen definiert. Mit fill="darkgoldenrod" wird die Füllfarbe des Histogramms festgelegt (nicht zu verwechseln mit color, welches die Farbe der Linien definiert).
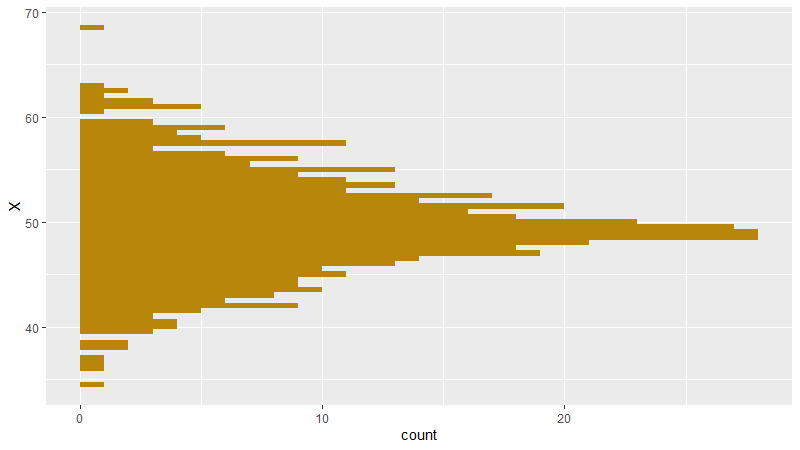
Titel und Achsen in ggplot2
Jetzt möchten wir das Histogramm noch einmal erstellen – mit einer Klassenbreite von 0.5, mit einer bläulichen Füllfarbe, einem minimalistisch schwarz-weißen Hintergrund und mit Titel sowie Achsenbeschriftungen. Here we go:
ggplot(dt, aes(X)) +
geom_histogram(binwidth=0.5, fill="steelblue") +
theme_bw() + ggtitle("Histogramm von X") +
xlab("Wert") + ylab("Häufigkeit")Die ersten zwei Teile kennen weir bereits. Hinzugekommen sind: theme_bw(), welches ein „black/white theme“ zur Folge hat; ggtitle(…), der Titel des Plots; xlab(…) und ylab(…), die Achsenbeschriftungen. Es gibt übrigens noch weitere themes, die ich weiter unten benutzen werde, aber nicht explizit vorstelle.
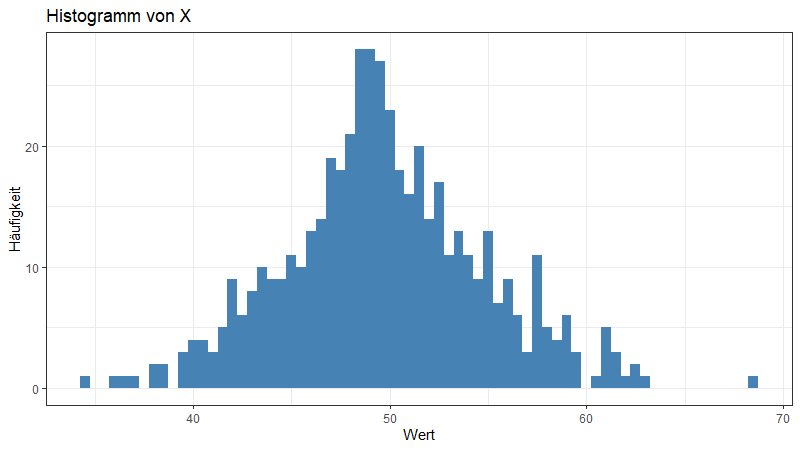
qplot in ggplot2
Bevor wir zu komplexeren Graphen übergehen, ein kurzer Hinweis: Mit der qlot-Funktion können wir ohne viel Schreibarbeit einen Plot erstellen, der das Nötigste mit den Standardeinstellungen enthält und an die base-R plot-Funktion angelehnt ist. Beispiel: qplot(X, data=dt)
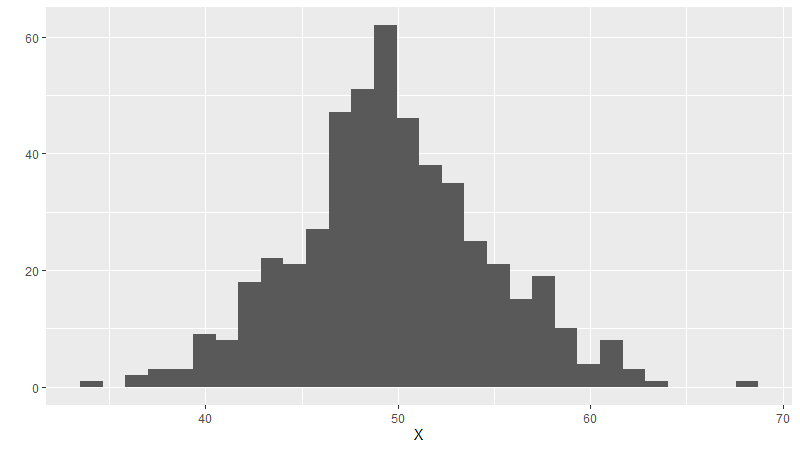
Dichtekurven mit ggplot2
Kommen wir von den Histogrammen weg und widmen uns den Dichtekurven. Diese lassen sich ganz einfach erstellen: ggplot(dt, aes(X)) + geom_density(fill="red", alpha=0.5) + theme_light(). Wie auch beim Histogramm gibt es eine Füllfarbe, ich habe hier noch ein weiteres Attribut alpha hinzugefügt, welches die Deckkraft der Farbe angibt (0 = vollständig transparent).
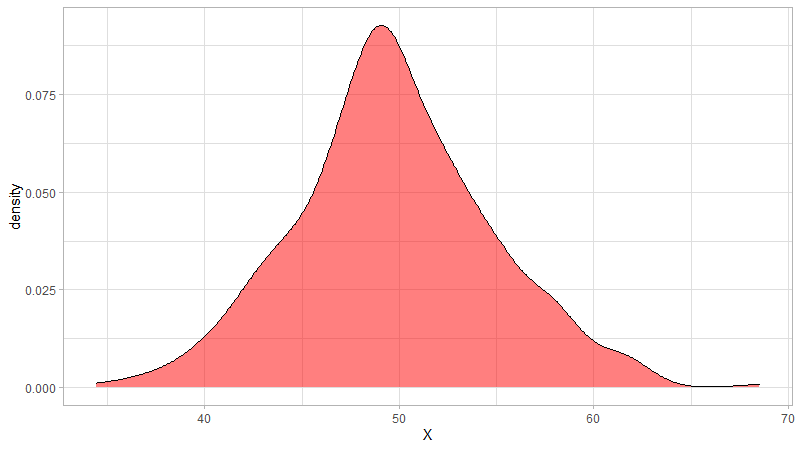
Zwei überlappende Dichtekurven
Wir möchten nun die Verteilungen von X und Y vergleichen. Ein logischer Schritt ist es, beide Verteilungen in einem Chart darzustellen. Ich zeige diesmal zuerst den Plot und gehe danach auf den Code ein:
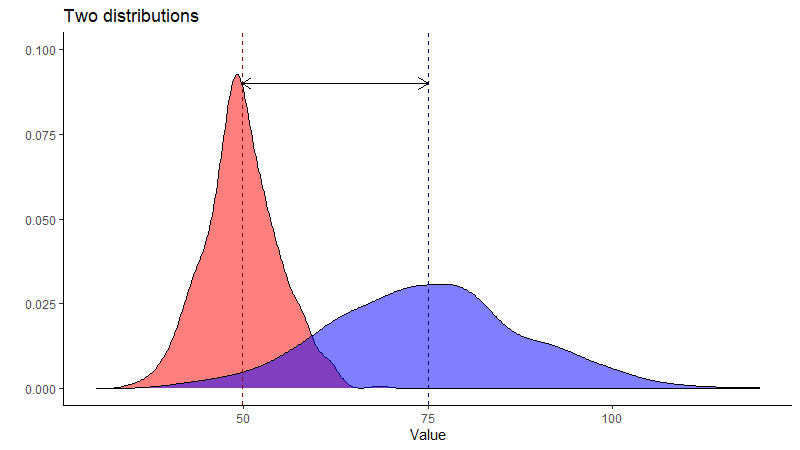
Der Code dazu sieht wie folgt aus:
ggplot(dt, aes(X)) + geom_density(fill="red", alpha=0.5) +
geom_density(aes(Y), fill="blue", alpha=0.5) +
geom_vline(linetype="dashed", color="darkred", xintercept=dt[, mean(X)]) +
geom_vline(linetype="dashed", color="darkblue", xintercept=dt[, mean(Y)]) +
geom_segment(data=dt, aes(x=mean(X), y=0.09, xend=mean(Y), yend=0.09),
arrow = arrow(length = unit(0.03, "npc"), ends="both")) +
xlim(c(30, 120)) + ylim(c(0, 0.1)) + theme_classic() +
ggtitle("Two distributions") + ylab("") + xlab("Value").
Nun, das ist etwas mehr Code als vorher. Schauen wir auf die wichtigsten Änderungen: Zum einen haben wir eine zweite geom_density, diesmal aber mit Y als Variable und einer anderen Füllfarbe. Mit geom_vline fügen wir eine vertikale Linie hinzu (Linientyp ist dashed). Am relevantesten ist hier das Attribut xintercept: Hier habe ich jeweils den Mittelwert der Variable übergeben. Zusätzlich haben wir noch einen Pfeil zwischen den Mittelwertslinien durch geom_segment; in aes(…) geben wir an, von wo bis wo der Pfeil gehen soll und mit arrow(…) werden die Eigenschaften des Pfeils festgelegt. Als letzte Neuerung gibt es noch die Befehle xlim und ylim – dies sind die „Limits“ der Achsen und können mit einem 2-Element-Vektor gesetzt werden (wichtig: es dürfen keine Daten abgeschnitten werden, die Limits müssen also immer größer sein als min und max der Daten).
ggplot2 und das Long Format
Im vorigen Beispiel hatten wir geom_density und geom_vline zwei Mal hintereinander benutzt. Hätten wir nun 5 Verteilungen, wäre es sehr unschön, fünf Mal nahezu den selben Code aufschreiben zu müssen. Und tatsächlich gibt es eine bessere Methode. Dazu müssen wir die Daten vom wide format ins long format transformieren. Hier ist ein Beispiel für ein wide format:
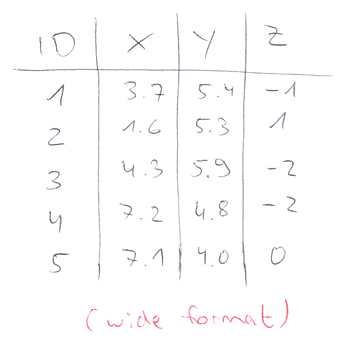
Diese Daten können wir ins long format bringen:

Wir erkennen schnell, woher die Benennung der Formate kommt.
Schön und gut, wie können wir das uns zunutze machen in ggplot? Dazu folgender Codeschnipsel:
dtLong <- melt(dt, id.vars="Group",
variable.name="Variable",
value.name="Value")
dtMeans <- dtLong[, list(Value=mean(Value)), by="Variable"]
ggplot(dtLong, aes(Value, fill=Variable)) + geom_density(alpha=0.5) +
geom_vline(data=dtMeans,
aes(xintercept=Value, color=Variable), linetype="dashed") +
geom_segment(data=dtMeans, aes(x=Value[1], y=0.09, xend=Value[2], yend=0.09),
arrow = arrow(length=unit(0.03, "npc"), ends="both")) +
xlim(c(30, 120)) + ylim(c(0, 0.1)) + theme_classic() +
ggtitle("Two distributions") + ylab("") + xlab("Value")Der ersten Befehl ist die Transformation. Bei data.table benutzen wir melt, um Daten vom wide ins long format zu bringen (benutze dcast, um Daten von long zu wide zu bringen). Anschließend erstellen wir eine abgeleitete Tabelle dtMeans, welche die Mittelwerte je Variable beinhaltet. Interessant wird die neue Art und Weise, den Plot zu erstellen. Eine Änderung ist der Grundbefehl ggplot(dtLong, aes(Value, fill=Variable)). Wir übergeben den transformierten Datensatz, als x fungiert hier die Spalte Value und die Füllfarbe ist nun abhängig von der Spalte Variable. Den Rest macht ggplot2 selbst. Das selbe Prinzip gilt für die beiden vertikalen Linien: hierfür haben wir dtMeans erstellt und das Mapping wird nun geändert auf aes(xintercept=Value, color=Variable). Dieser "long-Ansatz" ist häufig wesentlich eleganter und einfacher und Du solltest es immer erst hiermit versuchen.
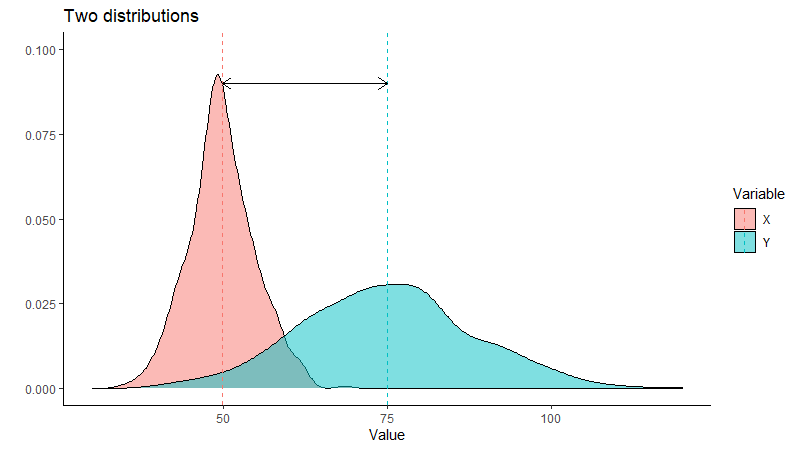
Aufteilen in mehrere Plots mit facet_wrap
Ein weiteres Extra, das mit dem long-Format sehr einfach funktioniert: Das automatische Erstellen von mehreren Plots je Gruppierung. Code:
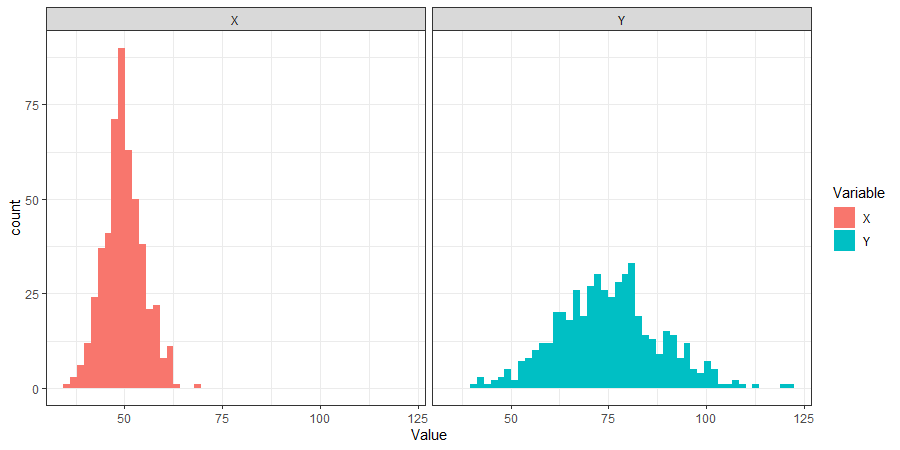
ggplot(dtLong, aes(Value, fill=Variable)) + geom_histogram(bins = 50) +
facet_wrap(~Variable) + theme_bw()Der relevante Teil ist hier das facet_wrap(~Variable). Hiermit wird gesagt, dass je Wert von Variable ein Plot erstellt werden soll.
Scatterplots mit ggplot2
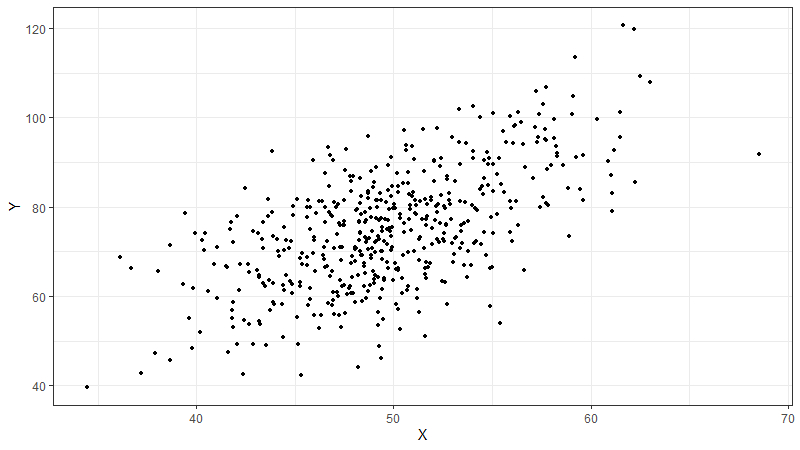
Ein Streudiagramm ("scatterplot") lässt sich mit ggplot2 auch ganz einfach erstellen. Hierzu verwenden wir lediglich geom_point als geometrisches Objekt: ggplot(dt, aes(X, Y)) + geom_point(size=1) + theme_bw(). Wir sehen: Es werden diesmal beide Variablen X und Y übergeben. Ebenso können wir die Größe der Punkte mit size als Attribut angeben.
Streudiagramm mit Regressionsgeraden
Wir können ganz einfach eine Regressionsgerade in den Plot mit einbauen:
ggplot(dt, aes(X, Y)) + geom_point(size=2) +
geom_smooth(method="lm", color="steelblue") + theme_bw()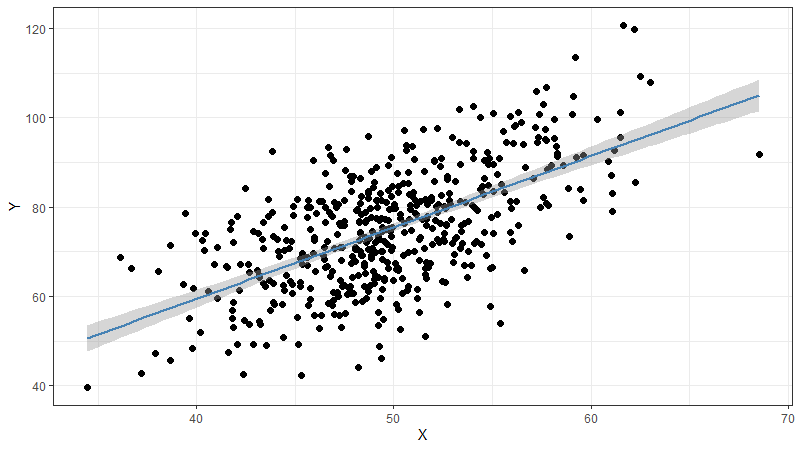
Wie zu sehen, benutzen wir dafür geom_smooth. Als method verwenden wir hier lm für ein lineares Modell; es wird standardmäßig von einer simplen linearen Regression ausgegangen (y = b0 + b1x). Auch sehen wir, dass hier ein Hinweis auf ein Fehlerintervall zusätzlich abgebildet wird (grauer Bereich). Dies lässt sich in geom_smooth mit se=FALSE auch ausschalten.
Verschiedene Regressionsgeraden je Gruppe
Können wir diesen Plot auch noch auf die Gruppen aufteilen (im Datensatz: Group-Variable)? Auch das ist ganz einfach möglich, indem wir es in aes angeben (wie bereits oben schon gemacht):
ggplot(dt, aes(X, Y, color=Group, shape=Group)) +
geom_point(size=1) + geom_smooth(method="lm", se=FALSE) +
theme_bw()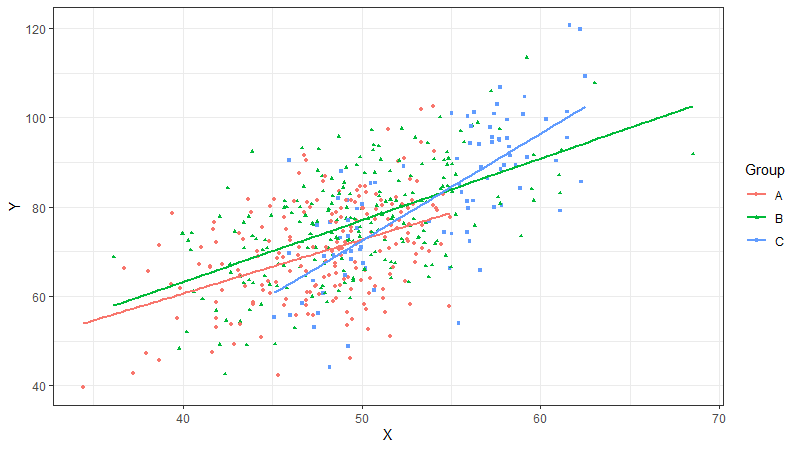
Ich habe hier gleich zwei visuelle Änderungen vorgenommen: Die Gruppe bestimmt sowohl die Farbe (color), als auch den Punktetyp (shape).
Boxplots in ggplot2
Da Du jetzt bereits die Details und Denke hinter ggplot kennst, werde ich mich hier kürzer fassen. Boxplots erstellst du mit geom_boxplot. In dem folgenden Beispiel sind es "side-by-side boxplots", also eine Darstellung je Gruppe.
ggplot(dt, aes(Group, Y, fill=Group)) +
geom_boxplot(alpha=0.5) + theme_bw()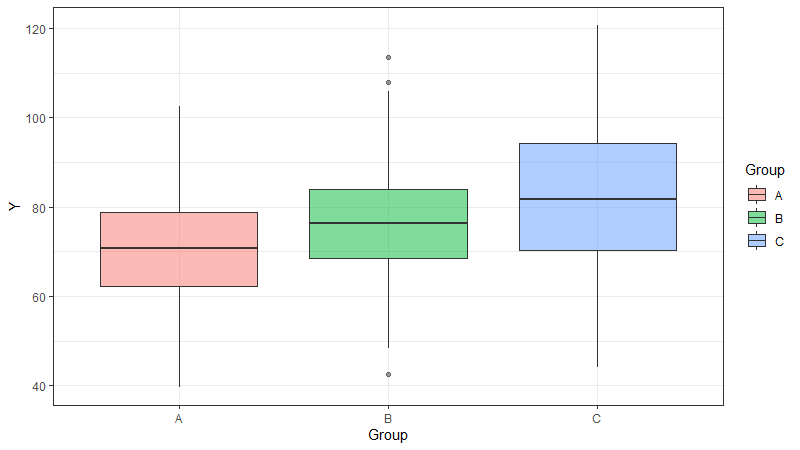
Violin Plots in ggplot2
Eine Abwandlung der bekannten Boxplots sind sog. "violin plots". Bei diesen kann man die Verteilung der Daten etwas besser erkennen.
ggplot(dt, aes(Group, X, fill=Group)) +
geom_violin(alpha=0.8) + theme_bw()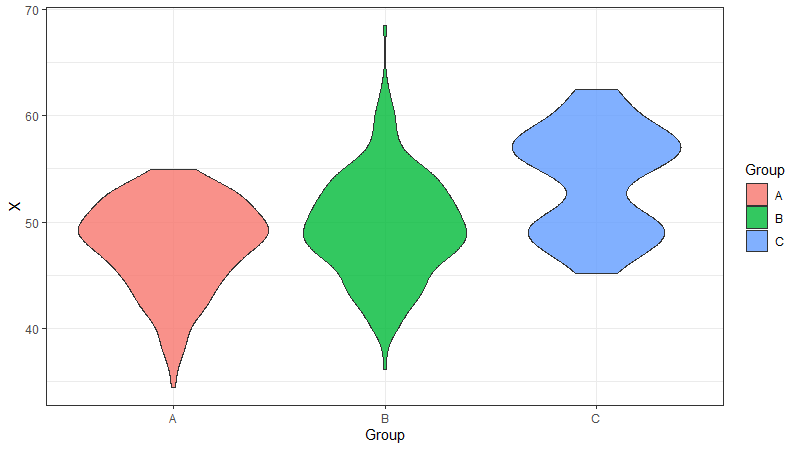
Error Bar Plots in ggplot2
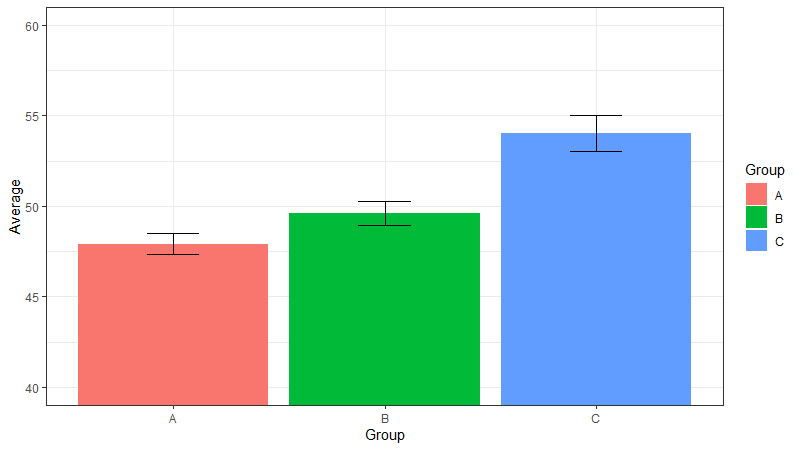
In Studien werden häufig Gruppen(mittelwerte) miteinander verglichen. Gute Graphen stellen hier nicht nur die Mittelwerte selbst, sondern auch die Standardfehler oder Konfidenzintervalle dar. In diesem Sinne habe ich hier einen solchen Plot vorbereitet, der die Mittelwerte sowie Konfidenzintervalle beinhaltet. Dazu müssen wir vorerst die Statistiken erstellen:
dtSummary <- dt[, list(Average = mean(X),
lower = mean(X)-1.96*sd(X)/sqrt(.N),
upper = mean(X)+1.96*sd(X)/sqrt(.N)),
by="Group"]Anschließend können wir diesen Datensatz benutzen, um den Plot zu erstellen:
ggplot(dtSummary, aes(Group, Average, fill=Group)) +
geom_bar(stat="identity") + coord_cartesian(ylim=c(40, 60)) +
geom_errorbar(aes(ymin=lower, ymax=upper), width=0.25) +
theme_bw()Erklärung: Die Balken entstehen durch geom_bar. Mit "identity" für das stat-Attribut geben wir an, dass die Daten (unsere Statistiken in dtSummary) bereits aggregiert sind und genau so übernommen werden können. geom_errorbar fügt die Fehlerbalken hinzu. Wozu das coord_cartesian? Dadurch können wir die Achsenlimits von der y-Achse einschränken, sodass die Balken abgeschnitten werden (wie oben bereits erwähnt ist das durch ylim nicht möglich). Kleiner Hinweis an der Stelle: immer vorsichtig sein mit dem Anpassen von Achsen! Die Art und Weise, wie man Daten/Ergebnisse darstellt kann schnell irreführend sein oder manipulativ wirken - noch ein Thema, das den Rahmen dieses Posts jetzt sprengen würde.
Zeitreihen mit ggplot2 darstellen
Zuletzt noch ein Beispiel, wie man Zeitreihen mit ggplot darstellen kann. In R gibt es verschiedene Möglichkeiten, Zeitreihendaten zu verwenden; hier werde ich weiterhin an der data.table-Struktur festhalten und benutze eine Date-Spalte für die "Zeit". Hier der Code für das Erstellen der Daten:
N <- 365
dtd <- data.table(
Date = as.Date("2020-01-01") + 1:N - 1,
Value = 50 + cumsum(rnorm(N, 0, 0.5) + rbinom(N, 1, 0.53) - 0.5)
)
fnChannel <- function(x, k=14, mode="min") {
len <- length(x)
res <- rep(NA_real_, len)
if (mode == "min") {
for (i in k:len)
res[i] <- min(x[(i-k+1):i])
} else if (mode == "max") {
for (i in k:len)
res[i] <- max(x[(i-k+1):i])
}
return(res)
}
dtd[, LowerChannel := fnChannel(Value, 14, "min")]
dtd[, UpperChannel := fnChannel(Value, 14, "max")]
dtd[Value==LowerChannel, HitLower := Value]
dtd[Value==UpperChannel, HitUpper := Value]Anschließend, der Code für den Plot:
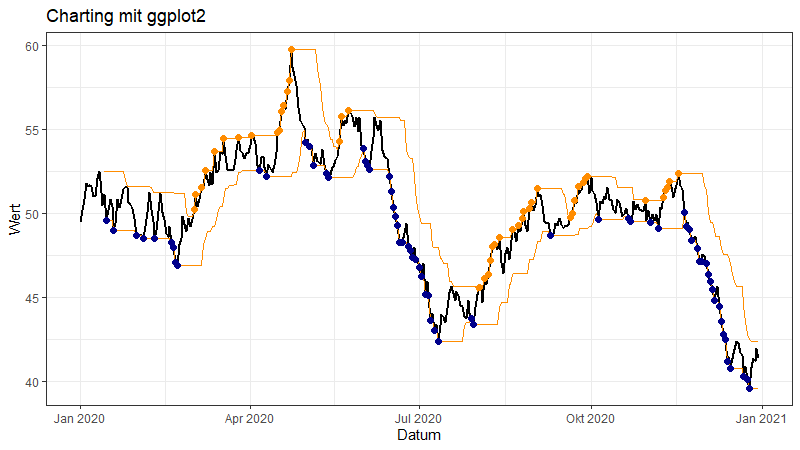
ggplot(dtd, aes(Date, Value)) + geom_line(size=1) + theme_bw() +
geom_line(aes(Date, LowerChannel), size=0.5, color="darkorange") +
geom_line(aes(Date, UpperChannel), size=0.5, color="darkorange") +
geom_point(aes(Date, HitLower), size=2, color="darkblue") +
geom_point(aes(Date, HitUpper), size=2, color="darkorange") +
ggtitle("Charting mit ggplot2") + xlab("Datum") + ylab("Wert")Folgender Plot entsteht (kleine Aufgabe: Wie könnte man das im Long-Format machen?):
Das war's nun auch mit meiner ggplot2-Einführung! Ich hoffe, der Post hat dir gefallen und dass ich dir helfen konnte. Frag' mich gern, wenn Dinge noch unklar sind oder Du weitere Aspekte lernen möchtest. Es hilft auch immer ein Blick in die Dokumentation von ggplot2, z.B. hier.
Viel Erfolg und bleib gesund!








2 Kommentare