Schleifen in R
Schleifen in R – das ist unser heutiges Thema. Wir behandeln for-Schleifen, while-Schleifen und schauen uns kurz zwei apply-Funktionen an, die häufig sehr hilfreich sind. Für einen Teil des Codes ist das data.table-Package notwendig. Für die Visualisierungen wird das ggplot2-Package benutzt.
Schleifen in R
Grundsätzlich wäre da erstmal die Frage: Was ist eine Schleife und wofür brauche ich diese? Schleifen sind Kontrollstrukturen (siehe if und else), die einen Codeblock wiederholen, solange eine Bedingung eingehalten wird. So wird üblicherweise zuerst geprüft, ob die gegebene Bedingung eintritt und dann – wenn dem so ist – der Codeblock einmal ausgeführt. Anschließend wird die Bedingung wieder geprüft. Dies geht solange, bis die Bedingung nicht mehr zutrifft (meistens sind es Programmierfehler, wenn es nie zu einem Abbruch kommt; das nennt man dann Endlosschleife).
Ein kurzes Beispiel vorweg
Fangen wir an und schauen auf folgenden Codeschnipsel: while (1 == 0) { print("Die Welt geht unter.") }. Wir haben eine while-Schleife, die zum Glück nie ausgeführt wird – weil die Bedingung nicht zutrifft. Schritt für Schritt: Das while signalisiert, dass wir hier eine while-Schleife haben; in den Klammern steht die Bedingung (1 ist gleich 0, was zu FALSE auflöst); dahinter steht ein Block {…}, der ausgeführt wird, sofern die Bedingung wahr ist – in diesem Fall also nie. Man kann die Schleife wie folgt lesen: „Solange eins gleich null ist, führe den print-Befehl aus“.
Im nächsten Absatz beschreibe ich zuerst for-Schleifen, da diese meiner Meinung nach häufiger vorkommen. Ich gehe später nochmal genauer auf while-Schleifen ein.
for-Schleifen in R
Schauen wir uns ein Beispiel an:
#Simple vector
fruitVec <- c("Apple", "Banana", "Orange", "Pear")
#Simple for loop
for (fruit in fruitVec) {
cat(paste0("The current fruit is: ", fruit, "\n"))
}Wir definieren einen Vektor mit Obstsorten. Nun möchten wir "durch diesen Vektor iterieren" und jedes Element einmal anzeigen lassen. Das können wir mit einer for-Schleife machen (zugegeben, es geht wesentlich einfacher, da R vektorisiert ist - die Auflösung gibt es später im Text). Das for ist das Schlüsselwort für die Schleife; fruit ist in diesem Beispiel der Platzhalter für das Element im jeweiligen Schleifendurchlauf, welches aus fruitVec stammt. Das Gerüst einer solchen Schleife ist also for (element in loopVector) { ... }.
for-Schleife mit seq_along
Es ist auch möglich, den Index / die aktuelle Position zu benutzen. Dies geht sehr gut mit der R-Funktion seq_along, welche einen Vektor mit Ganzzahlen von 1 bis N (Anzahl der Elemente im Vektor) zurückgibt:
#Another possibility
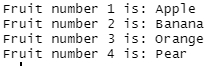
for (i in seq_along(fruitVec)) {
fruit <- fruitVec[i]
cat(paste0("Fruit number ", i, " is: ", fruit, "\n"))
}In dem Beispiel sieht man, wie wir i benutzen und uns so auch die jeweilige Obstsorte ermitteln (fruit <- fruitVec[i]).
Einfachere Methode
Wie versprochen, hier der Code, der das selbe Ergebnis produziert: cat(paste(paste0("Fruit number ", seq_along(fruitVec), " is: ", fruitVec), collapse="\n")). Es kann besser sein, for-Schleifen zu vermeiden und sich die Vektorisierung von R zunutze zu machen (dies kann performanter / eleganter sein).
while-Schleifen in R
Kommen wir nun zu den while-Schleifen. Das Beispiel oben lässt sich auch (komplizierter) mit einer solchen Schleife realisieren:
#Simple while loop
pos <- 1
while (pos <= length(fruitVec)) {
cat(paste0("Position ", pos, ": ", fruitVec[pos], "\n"))
pos <- pos+1
}Hier müssen wir eine zusätzliche Variable pos benutzen und selber darauf achten, diese im Schleifenblock zu erhöhen (pos <- pos+1).
Wann ist das Nutzen von while-Schleifen sinnvoll?
while-Schleifen sind dann sinnvoll, wenn wir eine Schleife erst dann verlassen möchten, wenn eine bestimmte Bedingung erreicht wird (und diese durch die Operationen in der Schleife beeinflusst wird). Ich habe dafür folgendes Beispiel geschrieben:
#Another while loop
value <- 0
resultVector <- value
counter <- 0
while (abs(value) < 100) {
counter <- counter + 1
value <- value + rnorm(1, 0, 2)
resultVector <- c(resultVector, value)
}
cat(paste0("It took ", counter, " iterations.\n"))
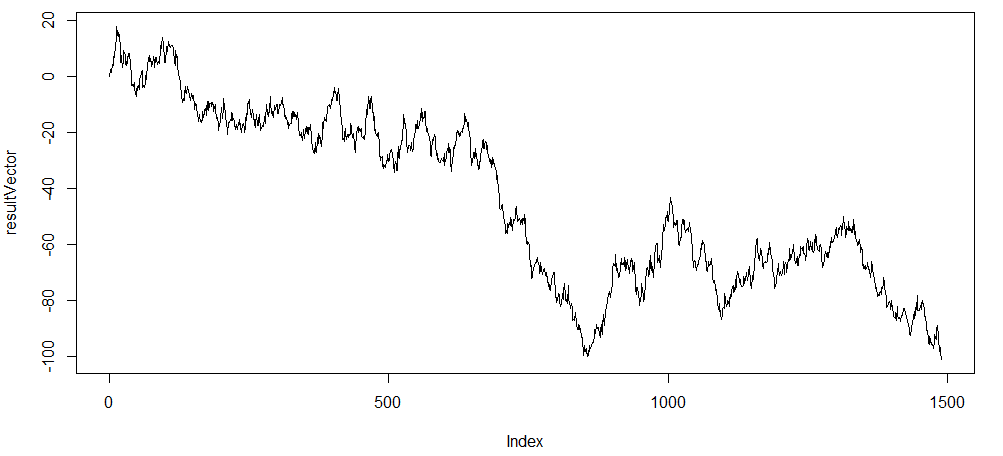
plot(resultVector, type="l")Hier passiert schon etwas mehr als vorher. Kurz gesagt haben wir einen Wert value, welcher mit einer Zufallszahl addiert wird. Diese Zufallszahl ist normalverteilt mit einem Mittelwert von 0 und einer Standardabweichung von 2 (rnorm(1, 0, 2)). Um den Verlauf des Prozesses im Nachhinein besser verfolgen zu können, speichern wir jeden Wert neu ab im resultVector. Die Schleife läuft solange, wie der Wert, also value, zwischen -100 und 100 liegt (= wie lange der absolute Wert abs(value) unter 100 ist). Mit der counter-Variable zählen wir die Anzahl der Iterationen.
Hier sehen wir den Verlauf von value:
Weitere Schleifen in R
Nun demonstriere ich weitere Fälle, in denen Schleifen genutzt werden können und werde später noch auf die apply-Funktionen eingehen, die R mitbringt.
Rollierender Median
Aufgabe: Schreibe eine Funktion, die für einen Vektor den rollierenden Median berechnet (z.B. Median aus den letzten 20 Fällen). Im folgenden Beispiel simulieren wir erneut Daten - eine Zeitreihe im Jahr 2020:
#Simulate timeline data
dateStart <- as.Date("2020-01-01")
dateEnd <- as.Date("2020-12-31")
dateRange <- seq(dateStart, dateEnd, by="1 day")
dt <- data.table(Date = dateRange)
dt[, Value := 25 + cumsum(rnorm(.N, 0, 2))]
#Plot
ggplot(dt, aes(Date, Value)) + geom_line(size=1) + theme_bw()Wir plotten die Zeitreihe mit ggplot, einer Funktion aus dem ggplot2-Package. Dieses solltest du auf jeden Fall parat haben, denn ggplot2 ist ein Must-Have für schöne R-Graphen.
Kommen wir nun zur Median-Funktion:
#Rolling median function
fnRollMedian <- function(x, period = 10) {
len <- length(x)
res <- rep(NA_real_, len)
for (i in period:len) {
posMin <- i-period+1
posMax <- i
res[i] <- median(x[posMin:posMax])
}
return(res)
}Was passiert hier? Zuerst wird die Startposition berechnet - diese ist period, denn wenn wir einen Median aus den letzten N (= period) Fällen berechnen möchten, dann können wir nicht bei 1 anfangen (es sei denn period ist 1...), sondern weiter oben im Vektor. In der for-Schleife iterieren wir also durch den Vektor period:len und in jedem Schritt berechnen wir den Median von dem Subvektor x[posMin:posMax]. Das Resultat schreiben wir in den Resultatsvektor res, den wir dann zum Schluss ausgeben.
Wir wenden die Funktion auf unsere simulierten Daten an; voilà!
#Compute rolling median
dt[, RollMed := fnRollMedian(Value, 20)]
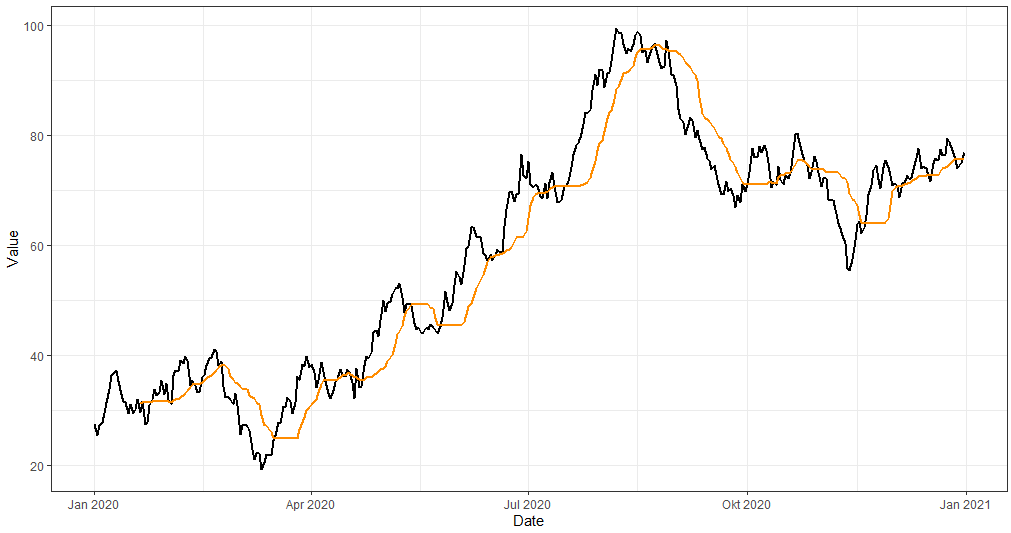
ggplot(dt, aes(Date, Value)) + geom_line(size=1) +
geom_line(aes(Date, RollMed), size=1, color="darkorange") +
theme_bw()Super - das sieht erwartungsgemäß aus:
Die apply-Funktionen in R
Zuletzt möchte ich kurz auf die apply-Funktionen aufmerksam machen, die eine Schleife häufig vereinfachen. Als Beispiel haben wir eine Liste mit Vektoren verschiedener Länge als Elemente:
mylist <- list(
SomeNumbers = 1:10,
SomeLetters = LETTERS[5:22],
SomeWords = c("Glass", "Laptop", "Moon", "Safari", "Social")
)Mit length(mylist) können wir lediglich die Anzahl der Elemente (eben die Länge der Liste selbst) herausfinden. Möchten wir aber die Länge der einzelnen Elemente berechnen, dann müssen wir durch die Liste iterieren. Statt nun z.B. eine for-Schleife zu schreiben, können wir es uns wesentlich einfacher machen: lapply(mylist, length). Wir bekommen mit diesem Befehl eine Liste zurück mit den Längen der Elemente. Es passiert folgendes: Das erste Argument von lapply ist in diesem Fall mylist, d.h. wir iterieren durch diese Liste. Je Element wird nun die Funktion angewendet, die als zweites Argument übergeben wurde, in diesem Falle length (aufpassen: hier ohne runde Klammern, da wir die Funktion als Objekt übergeben).
Übrigens: Häufig möchten wir einen Vektor erhalten (in diesem Falle einen Vektor mit den Längen der Elemente aus mylist). Dann können wir statt lapply ganz einfach sapply nutzen: sapply(mylist, length).
Das war's erstmal mit dem Thema Schleifen in R. Das waren die Basics, in einem Folgepost gehe ich auf next (sofort in den nächsten Schleifendurchlauf springen) und break (Schleife abbrechen) ein mit weiteren Beispielen. Außerdem gibt es noch einiges zu apply, z.B. Schleifen in Matrizen oder data.tables.
Falls dich ein bestimmtes Thema interessiert oder ich Code zu einer bestimmten Fragestellung präsentieren kann, sag gerne Bescheid!







Hallo, ich finde Ihren Beitrag sehr schön geschrieben. Falls Sie eine Fortführung machen, hätte ich einen Vorschlag – dieses Problem bekomme ich leider nicht gelöst… Ich möchte für einige Reihen (sagen wir A,B,C,…) diverse Befehle ausführen, wofür ich einen „Platzhalter“ bräuchte. Diese Programmierung gibt es aber meines Wissen nicht in R (in EViews würde das gehen). Also kommen hier auch Schleifen (oder apply) in Frage. Konkret möchte ich die Reihen zuerst in das ts-Format umwandeln, dann soll eine Schätung erfolgen (mit ETS), diese sollen dargestellt werden. Dann sollen forecasts gemacht werden und diese gespeichert (s. Befehle unten). Gibt es eine Möglichkeit, dies mit Schleifen durchzuführen? Falls Sie Ihren Beitrag erweitern, würde ich mich über einen Hinweis sehr freuen! Recht herzlichen Dank und Gruß, Michael Werner
#Umwandlung in Zeitreihenformat:
ReiheA <- ts(data$ReiheA, start=startdate, frequency=12)
ReiheB <- …
…
#Schätzung mit ETS-Methode:
ReiheA_fit <- ets(ReiheA)
ReiheB_fit <- …
…
#Darstellung der Schätzung:
ReiheA_fit
ReiheB_fit
…
#Forecast:
ReiheA_fore <- forecast.ets(ReiheA_fit, h=12)
ReiheB_fore <- …
…
#Speichern der Prognosewerte:
ReiheA_forevalue <- ReiheA_fore$mean
ReiheB_forevalue <- …
…
Hallo Herr Werner,
vielen Dank für ihr Feedback – solch eine Rückmeldung freut mich natürlich. Unter https://r-coding.de/blog/dynamisch-durch-spalten-iterieren/ habe ich eine Antwort veröffentlicht. Mich würde interessieren, ob der Code Ihnen ausreichend weiterhilft.
Beste Grüße
Hallo Thore,
das ist genau was ich gesucht habe. Super – vielen Dank! Da habe ich was gelernt, das ich auch in Zukunft gut gebrauchen kann!
Viele Grüße und schöne Feiertage,
Michael